免疫系统常识
western blot
免疫荧光
常规DNA的复制
常规DNA的复制如下图:【半保留复制】(semiconservative replication)
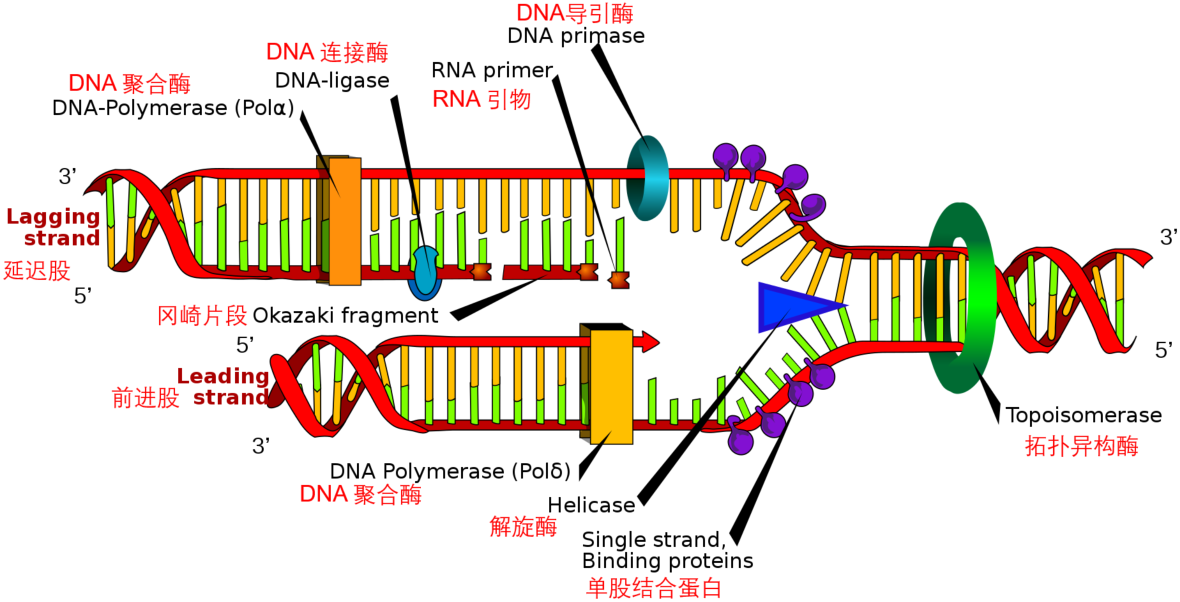
【解旋酶】(Helicase)
【拓扑异构酶】(Topoisomerase)
【导引酶】(Primase,也叫引物酶、引子酶)
【逆转录酶】(Reverse transcriptase)
【聚合酶】(polymerase):主要是连接DNA片段与单个脱氧核苷酸之间的磷酸二酯键, 在DNA复制中起做用
【Taq聚合酶】(Taq polymerase)
【连接酶】(ligase):主要是连接DNA片段之间的磷酸二酯键, 起连接作用, 在基因工程中起作用
【限制酶】(Restriction enzyme)
RT-PCR
实时荧光定量RT-PCR方法,核酸检测大体可以分为四个步骤:
第一步:提纯核酸
冠状病毒有蛋白质外壳、刺突、内部遗传物质(RNA,或者叫作核糖核酸)。采用裂解的方法,加入一些溶液,把外边的蛋白质外壳融掉,然后进行洗涤、离心等等,将RNA富集起来,得到比较纯的RNA。注意用棉签捅了嗓子眼之后,其实得到的主要是嗓子这个地方的上皮细胞,因此人体的DNA和RNA也会混杂在里面;另外如果这个人感染了像感冒这样的病毒,其DNA或者RNA也会在里面。有这么多RNA或者DNA,如何知道这里面有没有新冠病毒的RNA呢?
第二步:逆转录
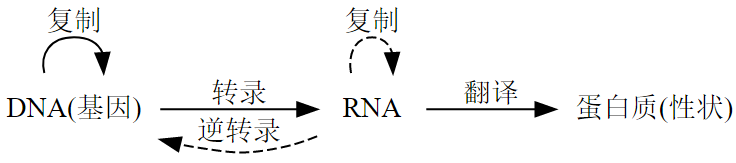
【中心法则】(Central dogma of molecular biology):以DNA (脱氧核糖核酸,A-T/C-G)为模板,根据碱基互补配对原则,合出一个单链的RNA (A-U/C-G),即转录,然后此RNA进入核糖体,翻译DNA的遗传密码,指导蛋白质的生产。
 类比:
类比:
- DNA是厂长,制定生产计划;
- RNA是车间主任,厂长告诉他生产什么,他就生产什么;
- 车间主任来到车间(核糖体),告诉核糖体怎么把这些氨基酸组成蛋白质。
注:
(1) 中心法则是由DNA双螺旋结构的发现者之一克里克于1958年提出,并且于1970年在Nature的一篇文章中重申:The central dogma of molecular biology deals with the detailed residue-by-residue transfer of sequential information. It states that such information cannot be transferred from protein to either protein or nucleic acid. (分子生物学的中心法则旨在详细说明连串信息的逐字传送。它指出遗传信息不能由蛋白质转移到蛋白质或核酸之中。换句话说,遗传信息传到蛋白质后,不能回流到核酸之中。) 【朊病毒】是目前已知的唯一例外。
(2) 为什么中心法则central dogma以dogma命名而不是law、rule等词?
【逆转录】(reverse transcription,缩写RT):RNA单链可以逆转录得到DNA。比如HIV病毒的遗传物质是RNA,可以转录为DNA,并且接到人体的DNA链上。
RNA是不太稳定的,容易坏掉;但是DNA是非常稳定的。既然如此,我们想找到病毒的RNA不好找,那么我们可以先将RNA逆转录为cDNA,那么我们可以来鉴定样本里面有没有cDNA来反推原来的样本里面有没有病毒的RNA。
第三步:PCR扩增
【PCR】(聚合酶链式反应,Polymerase chain reaction):是一项利用DNA双链复制的原理,在生物体外复制特定DNA片段的核酸合成技术。透过这项技术,可在短时间内大量扩增目的基因(标的基因),而不必依赖大肠杆菌或酵母菌等生物体。
生物学的两个时代:有PCR之前和之后。
PCR的三个步骤:
- 高温变性(Denaturation):93~98 ℃的高温使得DNA解旋,保温1~2 min;
- 低温退火(Annealing):50~65 ℃处理20~40 s,让【引物】(Primer)粘到DNA链上,此处的引物不来自引物酶,而是我们预先设计好的;
- 中温延伸(Extension/elongation):75~80 ℃处理,有了引物粘到DNA上之后,周围如果有核苷酸,这些核苷酸就会被引物拉着一个个排列到DNA上进行复制,每复制1000 个核苷酸大约需要1 min,该过程相当于传统复制中聚合这个步骤。在这个过程中,因为每个链的引物只有一个,和自然界中多个引物是不同的,因此不需要DNA连接酶,不会形成【冈崎片段】,但是还是需要DNA聚合酶,把周围的核苷酸给拽过来。
按照上面的步骤,循环40次,那么一个DNA就会变成240个,大概是1万亿个,即1万亿倍的扩增。上面的步骤中存在一个问题,即一般的酶不耐高温,那种第三步中的聚合酶会在下一次Denaturation过程中变性失效,如果每次循环都去添加聚合酶的话,又会很繁琐,那么能不能找打一个能耐高温的聚合酶呢? 1976年辛辛那提大学的研究生钱嘉韵在美国黄石公园的一种嗜热菌上提取出满足此要求的聚合酶,即【Taq聚合酶】,用于PCR扩增,大大便利了循环实验的操作。
【引物的特异性】(Primer's specificity):引物本身就只与目标DNA的某个片段进行互补。要实现这个功能,首先你必须知道病毒的DNA或者RNA的碱基序列,即需要测序。
第四步:实时荧光定量检测
前面的三个步骤我们已经有选择地让目标病毒的RNA变成DNA然后扩增,那么我们如何知道这里面的DNA是否真的进行了扩增呢?传统的方法是进行【电泳】(Electrophoresis),这个过程非常复杂。后来人们有发明了一种可以实时检测的方法——实时荧光法,让这个过程大大地加速;即通过荧光的信息来判断这里面有没有病毒,以及病毒的含量。
首先有一个荧光探针,现在比较流行的荧光探针是【TaqMan探针】,即在一段核苷酸链的两端分别连接Fluorophore(荧光基团)和Quencher(猝灭基团),该核苷酸链可以和目标碱基进行互补,设计的时候可以让其只能粘附在我们想要的DNA链上。探针的荧光基团和猝灭基团靠得比较近,所以不会发荧光;但是如果探针的核苷酸链粘附在目标DNA链上,而引物也粘附在目标DNA链上,并且从该引物处开始,在DNA聚合酶的帮助下,不断向两侧延伸DNA直到和探针的核苷酸链相遇,此时DNA聚合酶就会发挥其剪刀的作用,即把荧光基团给剪掉了,此时用激发光照射,该荧光基团就会发光。荧光越强,说明复制的目标DNA越多。
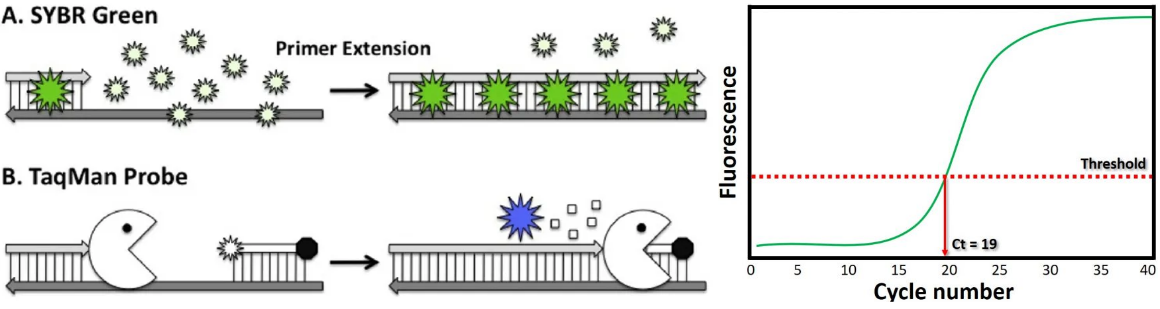
上图还给出了【SYBR Green】的方法,SYBR Green是一种染料,该染料优先与双链 DNA 结合,但对单DNA 的染色性能较低,染色后,在蓝光照射下可发绿光,因此可以用作双链DNA定量的染料。
有多种多样的Optical Detection System来检测荧光(参考Components of Real-Time PCR Detection Systems)。Light sources可以采用LEDs、Halogen lamp、Laser。Detectors可以采用photodiode、CCD、PMT。下面几个图分别对应CFX96 Touch optics shuttle、MiniOpticon™ real-time PCR detection system optics、iQ™5 real-time PCR detection system optics以及参考文献。

参考资料:
(1) 核酸检测原理是什么?如何从千万人中找到阳性感染者?—李永乐
(2) 如何才能长生不老?生命的时钟在哪里?—李永乐
(3) DNA 中的 3' 端、 5' 端是什么意思?
(4) Real-time PCR: Principles and Applications
(5) gitbook